-
解析応用編
2. マクネマー検定 McNemar test
このサイトは無料の統計ソフトである「R」を用いて
マクネマー検定 McNemar test
を実行できるように説明したサイトです。
「臨床医のためのRコマンダーによる医学統計解析マニュアル」の読者を対象に作成しており、本書の内容を理解しているものとして解説していきます。
その他、臨床研究や英語論文執筆にご興味のある方は無料のメールマガジン
「臨床研究の立ち上げから英語論文発表までを最速最短で行うための極意」への登録も御考慮下さい。
マクネマー検定は対応のある場合の比率の差の検定です。
カイ二乗検定のペアを考慮した場合の検定だとも言えます。
検定にはmcnemar.test( )関数を使用し、Table形式のデータに関数を使用することが可能ですが、Table形式のデータを作成するに当たって少しデータの型を調整してあげる必要があります。
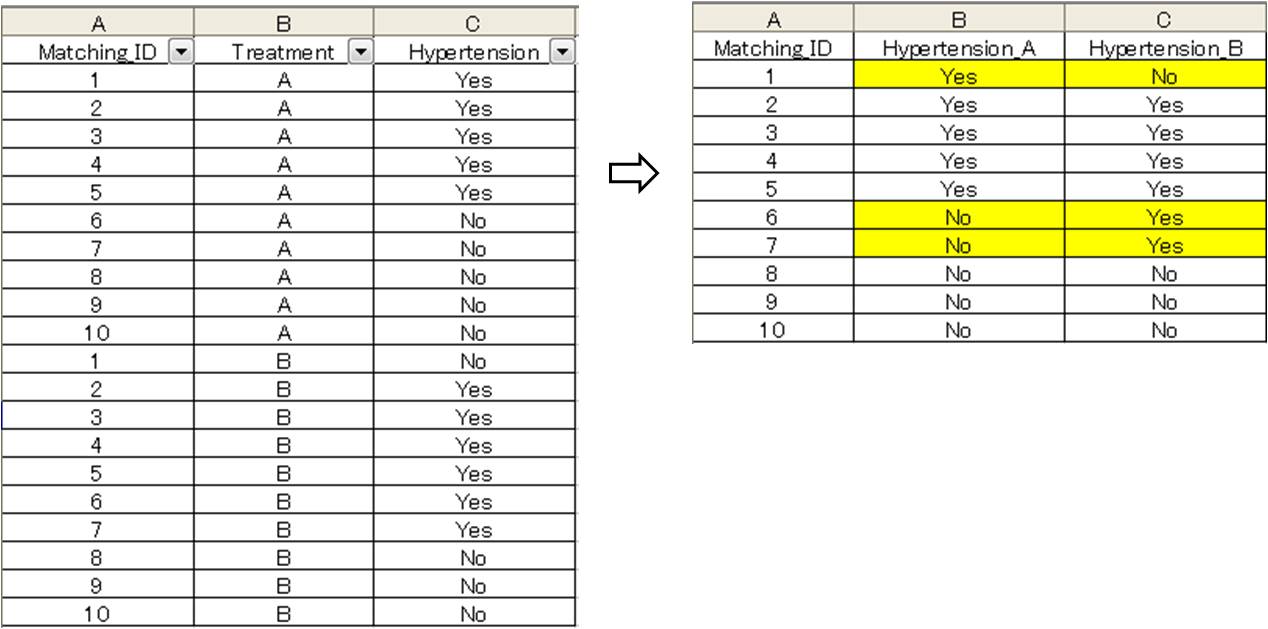
下図左は、ペアを考慮したデモデータになります。
ペアの相手をMatching_ID、治療内容をTreatment、高血圧症の有無をHypertensionの列に記載しており、Matching_IDとTreatmentでソートしています。しかしこのままではマクネマー検定を行う事ができませんので、このデータを下図右のような形に修正して下さい。すなわち、高血圧症の有病率を治療A群と治療B群でペアを考慮して検定するためには、ペアである事がわかるようにMatching_IDで行を揃えて、治療A群の高血圧症の有無、治療B群の高血圧症の有無の情報の列を作成する必要があります。
次に右の型のデータをRコマンダーで読み込みます。データ名は「Dataset」とします。
Rコマンダーで「統計量」→「分割表」→「2元表」から仮説検定のチェックをはずして解析を実行して下さい。
下記のようなスクリプトがスクリプトウィンドウに表示されます。
.Table <- xtabs(~Hypertension_A+Hypertension_B, data=Dataset, subset=)
.Table
remove(.Table)
マクネマー検定を行うにはここでremove(.Table)を削除して
mcnemar.test(.Table, correct=FALSE)を追記して下さい。下記のような状態になったらスクリプトをドラッグ&ドロップで選択し「実行」をクリックして下さい。以上でマクネマー検定が実行できます。
.Table <- xtabs(~Hypertension_A+Hypertension_B, data=Dataset, subset=)
.Table
mcnemar.test(.Table, correct=FALSE)
出力ウィンドウには以下のような結果が得られます。
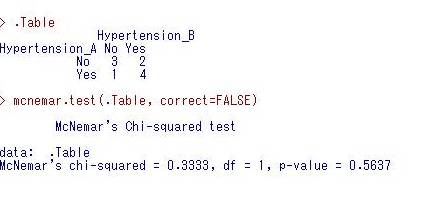
すなわち、ペアを考慮した場合
治療群AでHypertensionがYesで治療群BでもYesだった人が4人
治療群AでHypertensionがNoで治療群BでもNoだった人が3人
治療群AでHypertensionがYesで治療群BではNoだった人が1人
治療群AでHypertensionがNoで治療群BではYesだった人が2人
であり、McNemar検定の結果p=0.5637と、両群間で高血圧症の有病率に有意な差は認めなかったというように解釈します。
なお、このデータの加工が面倒で分布の数だけ計算したいという場合には以下のスクリプトを数値の部分のみ修正してコピー&ペーストするだけでマクネマー検定を実行することが可能です。
Table_Data <-matrix(c(4, 2, 1, 3), nrow = 2,
dimnames = list("1st Survey" = c("Yes", "No"), "2nd
Survey" = c("Yes", "No")))
Table_Data
mcnemar.test(Table_Data, correct=FALSE)